Virus
 Traduction
Traduction
Virus (Français)
/ Concept en Anglais (the virus)
/ Concept en Arabe (الفيروسات)
/ Concept en Italien (virus)
/ Concept en Russe (вирусы)
/ Concept en Chinois (病毒)
/ Concept en Suédois (virus)
/ Concept en Luxembourgeois (Virussen)
 Définition
Définition
Domaine, Discipline, Thématique
Définition écrite
Modèle:Les virus sont des parasites de tous les types de vie cellulaire car ils ne peuvent pas se reproduire. Ils infectent une cellule hôte et utilise sa machinerie pour produire d'autres particules de virus.
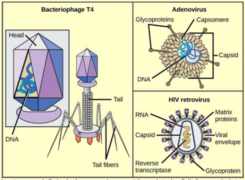
La particule virale infectieuse, appelée virion, est constituée d'un acide nucléique (ADN ou ARN) entouré d'une enveloppe protéique, la capside, constituée de nombreux exemplaires d'un ou plusieurs types de protéines. Les virus sont des parasites de tous les types de vie cellulaire car ils ne peuvent pas se reproduire. Ils infectent une cellule hôte et utilise sa machinerie pour produire d'autres particules de virus.
La particule virale infectieuse, appelée virion, est constituée d'un acide nucléique (ADN ou ARN) entouré d'une enveloppe protéique, la capside, constituée de nombreux exemplaires d'un ou plusieurs types de protéines. Les virus représentent la forme de vie la plus abondante et la plus diverse sur Terre. Ils peuvent infecter tous types d'organismes (vertébrés et invertébrés, plantes, champignons, bactéries, archées).
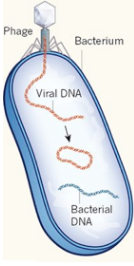
Les virus qui infectent les bactéries sont appelés bactériophages ou phages : ils représentent la forme biologique la plus abondante dans la biosphère avec environ 4,8 1031 phages (10 fois plus nombreux que les bactéries) et une biomasse estimée à 200 millions de tonnes.
Les océans (environ 70% de la surface de la Terre) représentent le principal réservoir de virus : chaque mL d'eau de mer contient environ 107 - 108 phages et environ 1023 infections par des phages se produisent chaque seconde. Les virus des archées jouent un rôle majeur dans la formation des sédiments océaniques en tuant leurs hôtes : cela entraîne la libération de 3 à 5 108 tonnes de carbone par an. L'intestin des mammifères est également densément peuplé : 1013 - 1014 bactéries et jusqu'à 108 particules virales (la plupart étant des bactériophages) par gramme de matière fécale (les phages d'Escherichia coli dominant : 103 - 104 phages chez l'homme et jusqu'à 107 chez la vache et le porc). Il est admis que les virus sont polyphylétiques et différents scénarios quant à leur origine sont envisagés :
Une évolution à partir d'éléments génétiques primordiaux pré-cellulaires. A l'inverse, une régression d'ancêtres cellulaires. La "fuite" de gènes d'hôtes d'origine qui auraient acquis une autonomie de réplication partielle et auraient été transformés en éléments génétiques parasitaires. L'étude des virus de plantes est à l'origine des premières expériences de biologie moléculaire. En 1935, Wendell Stanley (co-lauréat avec John Howard Northrop du Prix Nobel de chimie en 1946 "for their preparation of enzymes and virus proteins in a pure form") a purifié et partiellement cristallisé le virus de la mosaïque du tabac (dont il attaque les feuilles).
Infections virales médiatisées : virus du chikungunya (moustique "tigre" Aedes albopictus ou Aedes aegypti), virus de la dengue (moustiques du genre Aedes), virus HIV-1 (sida), virus Zika (Flavivirus, moustiques du genre Aedes).
exemple
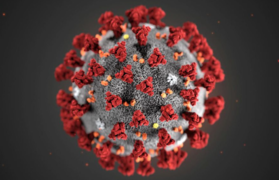
Les virus de la grippe
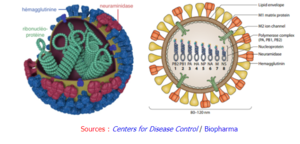
Il existe 4 types de virus de la grippe : A, B, C et D. Le type A est celui dont l'infection est la plus virulente chez l'homme.
Ce sont des virus à ARN qui codent jusqu'à 11 protéines dont l'hémagglutinine (HA) et la neuramidase (NA) : ces protéines ont une structure semblable à des pointes ("spike-like proteins") à la surface du virus (figure de gauche ci-dessous). Les sous-types du virus de la grippe A correspondent à 18 variants de HA et 11 variants de NA (figure de droite ci-dessous) : les nombres H et N indiquent la souche du virus (exemples : H1N1, H5N1 ou virus de la grippe aviaire).
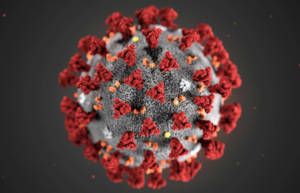
Le coronavirus SARS-CoV-2 ("Severe Acute Respiratory Syndrome COronaVirus 2", francisé en SRAS-CoV-2 (Syndrome Respiratoire Aigu Sévère), initialement 2019-nCoV puis "SARS-CoV-2 isolate Wuhan-Hu-1 genome". SARS-CoV-2 utilise une ARN polymérase ARN dépendante pour la réplication de son génome et la transcription de ses gènes. Ce virus est responsable de la maladie Covid-19 ("Co" : corona, "vi" : virus, "d" : disease, "19" : 2019). Figure ci-dessous : particules virales libérées de la surface d'une cellule infectée par le coronavirus SARS-CoV-2
Source de l'image : Nicholls et al., Université de Hong Kong
MERS-CoV : "Middle East Respiratory Syndrome COronaVirus" ou coronavirus du syndrome respiratoire du Moyen-Orient.
SADS-CoV : "Swine Acute Diarrhoea Syndrome COronaVirus" ou coronavirus du syndrome de diarrhée aiguë porcine. Quatre autres coronavirus humains (HCoV-NL63, HCoV-229E, HCoV-OC43 et HKU1) induisent des maladies respiratoires supérieures légères chez des hôtes immunocompétents.
Quelques définitions
Phage : un virus qui infecte un procaryote (bactérie et archée). Bactériophage : un virus qui infecte les bactéries. Exemples : phage λ, T2, T4, φ174, MV-11 Prophage : nom donné au génome d'un bactériophage quand il est intégré dans le chromosome de la bactérie hôte ou inséré dans cette batérie sous forme d'un plasmide extrachromosomique. C'est une forme latente du bactériophage qui ne perturbe pas la bactérie (phase lysogène)
Virophage : un virus qui infecte un autre virus. Exemple : le virophage Spoutnik est un virus satellite qui se reproduit dans des cellules infectées par un virus assistant. Spoutnik utilise le virus assistant comme hôte pour la reproduction et inhibe la réplication de ce dernier. Le virus assistant est Mamavirus, un grand virus (famille Mimiviridae). Virulence : elle peut être définie de différentes manières. La définition la plus simple est la sévérité ou la nocivité d'un agent pathogène. Virus enveloppé : virus entouré d'une bicouche lipidique dérivée de la membrane de la cellule hôte. Cette enveloppe protège le contenu du virion (voir ci-dessous) et contient, parmi d'autres protéines dérivées de la cellule hôte ou codées par le génome du virus, les protéines virales nécessaires à la fixation, à la fusion et à l'entrée du virus dans la cellule hôte. Récepteurs de la cellule hôte : ils sont la cible des glycoprotéines de fusion d'un virus enveloppé pour sa fixation spécifique à la cellule hôte et l'initiation de la cascade des évènements [fusion - entrée]. Virion : forme de transport d'un virus entre cellules. La maturation des virions a lieu après la sortie de la particule infectieuse virale de la cellule hôte soit par bourgeonnement soit par scellement de la capside sphérique. Cette maturation est un réarrangement et/ou un clivage irréversibles des protéines du virus qui activent le virion et le rendent compétent pour la réinfection.
Les virus n'ont pas de structure cellulaire et ne peuvent pas effectuer de réactions métaboliques. En revanche, ils utilisent la machinerie métabolique et génétique de la cellule hôte qu'ils infectent pour se multiplier.
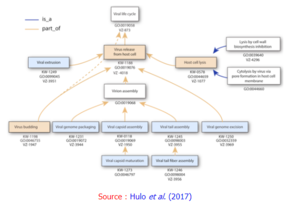
On peut les considérer comme des "parasites génétiques" et non comme des êtres vivants indépendants. Les caractéristiques structurales et fonctionnelles de leurs gènes sont semblables à celles des gènes de leurs cellules hôtes.
Contrairement aux génomes des autres êtres vivants, composés exclusivement d'ADN, certains génomes viraux sont constitués d'ARN. Certains virus sont dits à ARN négatif car leur matériel génétique est complémentaire de celui d'un ARN messager (qualifié de positif) et donc "illisible" par la cellule hôte.
Leur génome contient un nombre variable de gènes.
Exemple d'ontologie (termes et numéros d'accession GO, "Gene Ontology") des évènements clés du cycle de vie d'un virus (VZ : page de la base de données ViralZone; KW : mot clé UniProtKB)
Exemples de classification des virus - Les virus géants
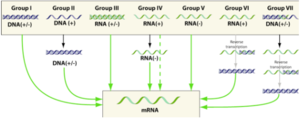
- Groupes des virus à ADN : groupe I = virus à ADN double brin ("double-stranded DNA virus" - "dsDNA"); groupe II = virus à ADN simple brin ("single-stranded DNA virus" - "ssDNA")
- Groupes des virus à ARN : groupe III = virus à ARN double brin; groupe IV = virus à ARN simple brin à polarité positive ("single-stranded RNA virus, positive-sense"); groupe V = virus à ARN simple brin à polarité négative ("single-stranded RNA virus, negative-sense")
- Groupes des virus (ADN ou ARN) à transcriptase inverse ("reverse transcriptase") : groupe VI = rétrovirus à ARN simple brin ("ssRNA(RT)"); groupe VII = rétrovirus à ADN double brin ("dsRNA(RT)")
Classification selon la méthode de David Baltimore sur la structure du génome :
- Group I : dsDNA ("double-stranded DNA") viruses; group II : ssDNA ("single-stranded DNA") viruses
- Group III : dsRNA ("double-stranded RNA") viruses
- Group IV : (+)sense RNA viruses; group V : (-)sense RNA viruses
- Group VI : positive-sense single-stranded RNA viruses that replicate through a DNA intermediate (retroviruses - reverse transcriptase to convert the positive-sense RNA into DNA).
- Group VII : double-stranded DNA viruses that replicate through a single-stranded RNA intermediate (pararetrovirus - reverse transcriptase)
David Baltimore a été co-lauréat du Prix Nobel de physiologie ou médecine en 1975 pour la découverte de la transcriptase inverse (enzyme des rétrovirus), conjointement à Renato Dulbecco et Howard Martin Temin "pour leurs découvertes concernant l'interaction entre les virus tumoraux et le matériel génétique de la cellule"
Classification de quelques familles de virus selon leur taille
- Geminivirus (15 - 20 nm) : génome à ADN, 2500 paires de bases.
- Phycodnavirus (110 nm) : génome à ADN double brin, 160.000 - 560.000 paires de bases.
- Iridovirus : (150 - 200 nm) : génome à ADN double brin, 100.000 - 220.000 paires de bases, environ 90 - 210 protéines prédites.
- Poxvirus (300 nm) : génome à ADN double brin, 130.000 - 300.000 paires de bases.
- Mimivirus (750 nm) : découverts en 2003 (Acanthamoeba polyphaga). Génome à ADN double brin (1,2 millions de paires de bases codant environ 900 protéines).
- Pandoravirus (1000 nm) : découverts en 2013. Ils possèdent un génome à ADN double brin avec la plus grande taille (2,5 millions de paires de bases, environ 2500 gènes) de tous les virus connus.
- Tupanvirus (Mimiviridae - 1200 nm) : découverts en 2018. Génome à ADN double brin (1,4 - 1,5 millions de paires de bases, 1276 à 1425 protéines prédites). Ces virus possèdent l'appareil de traduction le plus complet de la virosphère : en particulier, 70 ARN de transfert, 20 aminoacyl-tRNA synthétases, 11 facteurs pour les différentes étapes de la traduction, plusieurs facteurs de maturation des ARN de transfert et des ARN messagers .
- Pithovirus (1500 nm) : virus géant découvert en 2013. Il possède un chromosome circulaire à ADN double brin d'environ 610.000 paires de bases (467 gènes).
- Klothovirus (2500 nm) : virus géant mis en évidence en 2019 après ré-analyse de clichés anciens.
Les virus dits géants (découverts très récemment) ou virus à grand ADN nucléo-cytoplasmique ("NucleoCytoplasmic Large DNA Viruses", NCLDV) constituent l'un des plus grands groupes de virus qui infectent une grande variété d'hôtes eucaryotes. Exceptés les Poxvirus, les Pandoravirus et les Ascovirus, ils ont une forme approximativement icosaédrique. Les connaissances sur les NCLDV sont principalement dérivées d'isolats viraux co-cultivés avec des protistes et des algues. Cependant, 2074 génomes de NCLDV ont été reconstruits à partir d'échantillons prélevés à travers le monde et les données des métagénomes (Schulz et al., 2020).
- Le résultat est une augmentation de la diversité fonctionnelle et phylogénétique d'un facteur 10 et d'un facteur 11, respectivement.
- L'analyse de plus de 58.000 protéines majeures des capsides de grands virus et de virus géants à l'aide de données métagénomiques a permis d'établir un schéma de la distribution mondiale et de la nature cosmopolite de ces types de virus.
- Les génomes viraux découverts codent une large gamme de protéines avec des rôles potentiels dans la photosynthèse et divers processus de transport, ce qui indique que la reprogrammation de l'hôte est probablement une stratégie courante des NCLDV.
La diversité des NCLDV (qui sont par ailleurs associés aux principales lignées des eucaryotes) est en faveur de l'hypothèse qu'ils sont des acteurs importants des écosystèmes à travers les biomes.
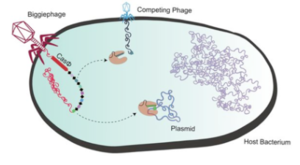
Les phages à très grand génome et leurs systèmes CRISPR-Cas
Les bactériophages ont généralement de petits génomes. Cependant, il existe des centaines de génomes de phages de plus de 200.000 paires de bases, jusqu'à 735.000 paires de bases. Dix clades de phages ont été identifiés et nommés : Mahaphage, Kabirphage, Dakhmphage, Jabbarphage, Kyodaiphage, Biggiephage, Whopperphage, Judaphage, Enormephage et Kaempephage (Al-Shayeb et al., 2020).
Les répertoires génétiques de ces phages à très grand génome contiennent divers systèmes CRISPR-Cas, des ARN de transfert (ARNt), des ARNt synthétases, des enzymes de modification de l'ARNt, des facteurs d'initiation et d'élongation de la traduction et des protéines ribosomales.
Les systèmes CRISPR-Cas de ces phages ont la capacité de réduire au silence les facteurs de transcription de la bactérie hôte et donc les gènes qui devraient être transcrits.
Les Biggiephages possèdent un système CRISPR-Cas très particulier :
Leur enzyme Cas (une seule protéine d'environ 70 kDa) a une taille environ la moitié de celles des autres systèmes CRISPR-Cas. Cette protéine Cas-Φ virale, plus petite et plus compacte, est donc plus facile à transporter dans les cellules pour éditer leur génome, car elles peuvent être emballées dans de petits systèmes de transport, notamment le virus désactivé appelé virus adéno-associé ("Adeno-Associated Virus" - AAV)
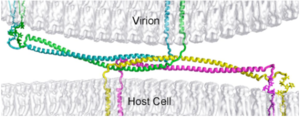
Entrée des virus enveloppés dans la cellule hôte
La reconnaissance et la fixation sur la cellule hôte est le préalable à toute infection par un virus. La membrane des cellules est caractérisée par de nombreux types de macromolécules (protéines, glycérophospholipides, glycanes, ...) dont le nombre, la structure et la composition sont la signature des cellules.
En conséquence, les nombreux virus ciblent ces macromolécules (qu'on appelle récepteurs) pour entrer dans la cellule hôte.
a.L'enveloppe virale est constituée par des éléments provenant de la membrane plasmique (bicouches lipidiques et protéines) de la cellule hôte ou du virus. Les virus remplacent cependant les protéines membranaires d'origine par leurs propres protéines, en particulier des glycoprotéines qui "pointent" et leur permettent de reconnaitre leurs cellules cibles et de se fixer à leur surface.
De nombreux virions sont donc recouverts de glycoprotéines ce qui leur confèrent l'aptitude à "communiquer" avec leur cellule hôte : le potentiel infectieux d'un virus dépend entièrement des glycoprotéines qui le caractérisent. De plus la membrane / enveloppe qui entoure les virus enveloppés leur permet d'entrer dans la cellule hôte par un mécanisme de fusion des membranes.
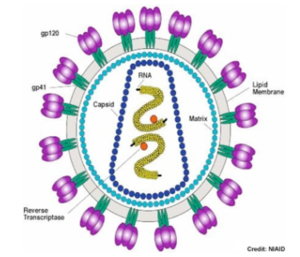
la glycoprotéine d'enveloppe Env du rétrovirus (lentivirus) HIV-1:
Cette glycoprotéine est synthétisée sous forme d'un précurseur appelé gp160 (PDB 4CC8) très flexible et recouvert de glucides. Ce précurseur est hydrolysé :
En une sous-unité gp120 exposée à la surface et impliquée dans la liaison au récepteur CD4 de la cellule hôte. En une sous-unité gp41 ancrée dans la membrane du virus et impliquée dans la fusion des membranes virale et cellulaire. Les sous-unités gp120 et gp41 s'associent de manière non covalente pour former un trimère d'hétérodimère [gp120-gp41]3. Du point de vue ultra-structural, elles forment des "pointes" dans l'enveloppe du virion.
Le trimère [gp120-gp41]3 du HIV-1 interagit avec le récepteur primaire CD4 et un corécepteur (tel que le récepteur de la chimiokine CCR5, un RCPG) pour fusionner les membranes du virus et de la cellule cible.
L'interaction entre gp120 et le corécepteur semble l'élément déclencheur de l'acquisition du potentiel de fusion de gp41.
Le récepteur CD4 ("Cluster of Differentiation 4") est une glycoprotéine présente à la surface de cellules immunitaires (cellules T auxiliaires, monocytes, macrophages, cellules dendritiques).
b. Incorporation des glycoprotéines dans l'enveloppe virale
L'appareil de synthèse des protéines de la cellule hôte ("producer cell") est utilisé pour synthétiser les glycoprotéines de la future enveloppe virale Env (figure ci-dessous). Ces glycoprotéines sont ensuite glycosylées et repliées correctement dans le réticulum endoplasmique de cette même cellule hôte. Elles sont enfin sécrétées (après transit par l'appareil de Golgi) et enchâssées dans la membrane de la cellule hôte. Elle sont alors prêtes à être recrutées par les nouvelles particules bourgeonnantes des virions en cours de formation. La synthèse de protéines virales dans la cellule hôte peut provoquer un stress dans le réticulum endoplasmique : les cellules hôtes disposent d'un mécanisme (appelé "Unfolded Protein Response" - UPR) pour répondre au stress lié à l'accumulation de protéines mal repliées dans le réticulum endoplasmique et ainsi l'atténuer.
Une particularité importante de l'UPR est sa capacité à augmenter la voie de dégradation des protéines associée au réticulum endoplasmique ("ER-Associated protein Degradation pathway" - ERAD), notamment via son mécanisme de contrôle de la qualité du repliement des protéines. C'est donc une "lutte évolutive" incessante entre les virus et la machinerie ERAD de leurs hôtes.
Modèles (non mutuellement exclusifs) d'incorporation des glycoprotéines Env dans l'enveloppe virale de nouveaux virions
Mécanisme 1 (figure ci-dessous) : les glycoprotéines Env à la surface de la cellule hôte pourraient être incorporées passivement dans les virions à mesure que les particules virales acquièrent une membrane dérivée de l'hôte pendant l'assemblage et le bourgeonnement.
lipidique", concentrant ainsi Env sur les sites d'assemblage.
Mécanisme 3 : des données expérimentales suggèrent que le domaine de la protéine Gag orienté vers la matrice et la queue de la sous-unité gp41 orientée vers le cytoplasme (voir ci-dessous) de la protéine Env interagissent, ce qui induit la rétention des complexes Env au niveau des sites de bourgeonnement et leur recrutement dans les virions.
Mécanisme 4 : cette interaction pourrait être indirecte par le biais d'une protéine de l'hôte reliant les deux protéines virales.
c-Protéines de fusion virales et fusion Les glycoprotéines assurent le processus dit de fusion entre l'enveloppe virale et les membranes des cellules hôtes. Les glycoprotéines de fusion virales sont en conséquence de plus en plus des cibles de médicaments antiviraux
Mécanisme de fusion [enveloppe du virus - membrane de l'hôte]: C'est la mise en contact intime de 2 bicouches membranaires distinctes suivie de leur fusion en une seule. Les peptides de fusion sont les séquences hydrophobes qui interagissent directement avec la membrane cible. Les étapes (quasiment identiques pour les 3 classes de protéines de fusion) sont les suivantes :
- apposition rapprochée
- hémifusion : étape au cours de laquelle seules de petites régions des monocouches externes des membranes en contact se confondent. Les feuillets internes des deux bicouches sont encore distincts et le contenu du virion n'a pas accès au cytoplasme de la cellule hôte
- petits pores de fusion puis grands pores de fusion
classe1, classe2 et classe3 sont les trois classes de glycoprotéines de fusion virales sont pour l'instant identifiées sur la base des similitudes de structures. ( voir le tableau dans la définition graphique) Les protéines adoptent des structures quaternaires très particulières :
La structure pré-épingle à cheveux ("pre-hairpin intermediate") est une conformation intermédiaire (étendue et métastable) de la protéine de fusion virale, juste avant qu'elle ne se replie dans la conformation stable après la fusion. La structure trimère d'épingles à cheveux ("trimer of hairpins") d. Les peptides de fusion Induit par un signal associé à l'entrée dans la cellule à infecter (exemples : liaison au [récepteur/co-récepteur], fixation de protons dans un endosome), les glycoprotéines de fusion subissent plusieurs changements de conformation. Un segment hydrophobe de la chaîne polypeptidique, appelé peptide de fusion (ou boucle de fusion) interagit avec la membrane de la cellule cible et cet intermédiaire de pontage rapproche les deux membranes (virus et cellule). Les peptides de fusion sont localisés de 3 manières dans la séquence polypeptidique :
- à l'extrémité N-terminale ou proche : la plupart des protéines de fusion de la classe I ont un peptide de fusion N-terminal généré par protéolyse
- boucle simple interne
- boucle bipartite interne
Caractéristiques de quelques peptides de fusion viraux de protéines de fusion de la classe I,IIet III:( voir les tableau dans la définition graphique) Exemple des peptides de fusion des protéines de fusion de la classe I
*En premier lieu, l'intermédiaire dit en pré-épingle à cheveux ("pre-hairpin intermediate") est formé pour ancrer les membranes virale et cellulaire via les domaines transmembranaire et le peptide de fusion, respectivement.
*La présence du peptide de fusion dans l'ectodomaine exposé à la phase aqueuse est une caractéristique commune à toutes les protéines de fusion virales et une nécessité absolue pour leur fonction fusogène. Il semble que la séquence hydrophobe du peptide de fusion (habituellement à l'extrémité N-terminale, ou à proximité, de la sous-unité fusogénique, voir le tableau ci-dessus) est impliquée dans la partition initiale de la protéine de fusion dans la membrane cible.
*Au cours de cette première étape, la formation (exemple, hémagglutinine IF) ou la complétion (exemple, protéine Env du VIH) d'un enroulement prolongé par des hélices N-terminales serait la force de propulsion qui provoque l'exposition et la translocation du peptide de fusion au voisinage immédiat de la membrane cible.
*La structure suivante est le trimère d'épingles à cheveux (à faible énergie) dans laquelle, les extrémités N- et C-terminales de l'ectodomaine se rejoignent : l'enroulement à l'extrémité N-terminale crée des sillons dans lesquels les hélices C-terminales se logent selon une orientation antiparallèle via des interactions hydrophobes.
*L'énergie libérée pendant la formation du trimère d'épingles à cheveux serait utilisée pour rapprocher les membranes et induire leur fusion.
e. Incidence du pH des endosomes sur la fusion des membranes Un pH bas est un élément déclencheur de la fusion pour certains virus (orthomyxovirus, rhabdovirus, alphavirus, flavivirus, bunyavirus et arénavirus). Ces virus pénètrent dans les cellules par endocytose et fusionnent avec des endosomes précoces (exemple, le virus VFS) ou tardifs (exemple, le virus de la grippe).
Un pH faible induit des réarrangements de la structure des 3 classes de protéines de fusion qui permettent de positionner le peptide de fusion afin qu'il puisse s'enfouir dans la membrane cible.
Pour les protéines de fusion de la classe I, cela implique la séparation des domaines de la tête globulaire qui fixent la sous-unité de fusion dans son état de pré-fusion. Par exemple, la glycoprotéine G attache le virus VSV aux récepteurs LDL de l'hôte, induisant une endocytose du virion dépendante de la clathrine. Dans l'endosome, le pH acide induit des modifications conformationnelles du trimère de la glycoprotéine qui déclenchent la fusion entre le virus et la membrane endosomale. Illustration du virus de la grippe
Dans le cas du virus de la grippe, la glycoprotéine de liaison au récepteur de la cellule cible est l'hémagglutinine (HA) :
C'est une protéine de fusion virale de la classe I. Elle représente 80% des protéines de l'enveloppe virale. Elle se lie aux molécules d'acide sialique (voir ci-dessous) constitutives du récepteur de la cellule cible ce qui induit l'internalisation du virion (par endocytose dépendante de la clathrine ou par une voie indépendante de la clathrine et de la cavéoline). HA est donc responsable de la fusion de l'enveloppe virale avec la membrane de l'endosome. HA est une protéine trimérique et chaque sous-unité est constituée de 2 domaines (HA1 et HA2 ) reliées par des ponts disulfures :
Le plus grand domaine HA1 (le plus distant de l'enveloppe virale) assure la liaison aux récepteurs. HA2 est un domaine plus petit. Il ancre HA dans l'enveloppe virale et il contient le peptide de fusion impliqué dans l'interaction entre les membranes. Le faible pH des endosomes active la fusion en facilitant les changements conformationnels irréversibles de la glycoprotéine :
Les structures de HA à pH neutre et au pH de fusion révèlent plusieurs formes distinctes de HA, en particulier une conformation de HA2 sous forme d'une bobine triple hélicoïdale (longueur 150 Å) qui établit un pont entre les membranes du virus et de l'endosome.
Entrée des virus enveloppés dans la cellule hôte
Les cellules de mammifères sont couvertes de protéines et de lipides glycosylés et les acides sialiques (exceptés les glycosaminoglycanes) sont les unités les plus externes des constituants glycosylés de la membrane plasmique. Les acides sialiques sont donc les récepteurs de plusieurs classes de virus (les rôles spécifiques de l'acide sialique et des autres récepteurs du virus dépendent du virus et du type de cellule). Les deux formes les plus courantes d'acide sialique reconnues par les virus qui ciblent des cellules de l'homme et de mammifères sont l'acide 5-N-acétylneuraminique (Neu5Ac) et l'acide 5-N-glycolylneuraminique (Neu5Gc). De manière générale, le terme acide sialique désigne les dérivés de l'acide neuraminique. La plupart des virus non enveloppés ne fusionnent pas avec la membrane plasmique car ils ne possèdent pas de revêtement sous forme d'une bicouche lipidique (à l'inverse des virus enveloppés) : ces virus transitent par des vésicules d'endosomes, par l'appareil de Golgi ou le réticulum endoplasmique. Ainsi, après s'être liés aux récepteurs à la surface de la cellule cible, les virus non enveloppés pénètrent dans les cellules hôtes par macropinocytose, par endocytose médiée par la clathrine, par endocytose médiée par cavéole (invagination) ou par une combinaison de ces modes d'entrée.
a. Modification subséquente de la membrane par les protéines de la capside des virus non enveloppés Après leur internalisation, les virus non enveloppés (exemples : Adenoviridae, Papillomaviridae, Polyomaviridae, Parvoviridae, ...) doivent [rompre / solubiliser / dégrader] :
La membrane plasmique (dans le cas des virus non enveloppés qui entrent par fusion des membranes comme les virus enveloppés). Ou la membrane de la vésicule ou du compartiment sub-cellulaire où ils se trouvent. La libération dans le cytosol de la nucléocapside de ce type de virus est généralement le fruit de peptides (de 10 à 60 résidus d'acides aminés*) de caractère global hydrophobe ou amphipathique. Ces peptides sont générés par protéolyse (autocatalytique ou par des protéases du virus ou de l'hôte) de polyprotéines précurseurs de la capside virale. Toute anomalie lors du clivage protéolytique réduit donc considérablement le pouvoir infectieux de ces virus. Ces peptides sont des molécules d'intérêt thérapeutique pour le transfert de gènes.
- Certains de ces peptides sont de plus longs fragments protéiques.
Le génome des picornavirus est un ARN simple brin de 7100 à 8900 paires de bases. Une protéine virale (VPg) est liée de manière covalente à l'extrémité 5' de l'ARN et une queue poly(A) en 3'. La région codante est flanquée de régions 5' et 3' non traduites (NTR). Illustration : structure du génome des picornavirus et maturation des polyprotéines par protéolyse
La longue région 5' non traduite contient un site d'entrée interne des ribosomes qui dirige la traduction des polyprotéines. Le cadre de lecture ouvert unique du génome est organisé en 1ABCD / 2ABC / 3ABCD, les numéros indiquant les 3 domaines différents et chaque lettre représentant une polyprotéine.
La région P1 code pour les protéines de structure de la capside. Les régions P2 et P3 codent pour les protéines non structurales associées à la réplication. Les polyprotéines de certains picornavirus possèdent une protéine supplémentaire, la protéine L. Ces polyprotéines sont des entités biologiques remarquables : elles possèdent toutes les propriétés (structurales et fonctionnelles) nécessaires à ces étapes du cycle de vie du virion. b. Mécanismes de perturbation des membranes pour la libération du contenu de la capside L'extrémité N-terminale de VP1 est un domaine phospholipase A2 (PLA2) : l'activité PLA2 réarrange la bicouche lipidique de l'endosome de manière transitoire et sur une échelle limitée. La protéine μ1 (76 kDa, protéine majeure de la capside des Réovirus de mammifères) est hydrolysée (autocatalytise) en deux fragments protéiques associés au virion : μ1N (4 kDa, myristoylé) et μ1C (72 kDa). La protéine μ1N forme des pores sélectifs dans la membrane endosomale. Il n'est pas certain que ces pores soient liés à la lyse osmotique de l'endosome, mais ils pourraient expliquer que le gros noyau de ce type de virus soit transfèré dans le cytoplasme.
- ......................................................................
.......................................................................
.......................................................................
....................................................................... ....................................................................... .......................................................................
....................................................................... ....................................................................... |
Définition graphique
 Concepts ou notions associés
Concepts ou notions associés
infection virale / parasite / cellule hôte / ARN / ADN / Bactériophage / génome / capside / multiplication virale /
 Exemples, applications, utilisations
Exemples, applications, utilisations
................................................................................ ................................................................................ ................................................................................
................................................................................ ................................................................................ ................................................................................ |
 Erreurs ou confusions éventuelles
Erreurs ou confusions éventuelles
- Confusion entre ....... - ........
- Confusion entre ....... - ........
- Erreur fréquente: ....................
 Questions possibles
Questions possibles
- Qu'est ce qu'un phage?
- C'est quoi un bactériophage?
- C'est quoi un virophage?
- Qu'est ce qu'il différencie un virus enveloppé?
- Quelles sont les constituants d'un enveloppe de virus?
- Comment se fait la synthèse des protéines virales dans la cellule hôte?
- Quel est le mécanisme de fusion: enveloppe du virus-membrane de l'hôte?
- quel est la Modification subséquente de la membrane de l'hôte par les protéines de la capside des virus non enveloppés?
- comment se fait l'intégration du génome viral?
- comment se fait la réplication et transcription du génome viral et la traduction des protéines virales?
- Quel est le mécanisme des médicaments antiviraux?
- ..................?
- ..................?
- ..................?
 Liaisons enseignements et programmes
Liaisons enseignements et programmes
Idées ou Réflexions liées à son enseignement
Aides et astuces
Education: Autres liens, sites ou portails
 Bibliographie
Bibliographie
Pour citer cette page: ([1])
ABROUGUI, M & al, 2021. Virus. In Didaquest [en ligne]. <http:www.didaquest.org/wiki/Virus>, consulté le 25, novembre, 2024
- ..................
- ..................
- ..................
- ..................
- Pages utilisant des arguments dupliqués dans les appels de modèle
- Sponsors Education
- Santé (Concepts)
- Microbiologie (Concepts)
- Hygiène (Concepts)
- Immunologie (Concepts)
- Médecine (Concepts)
- Biologie cellulaire (Concepts)
- Génétique (Concepts)
- Education à la santé (Concepts)
- Epistémologie (Concepts)
- Infection virale
- Parasite
- Cellule hôte
- ARN
- ADN
- Bactériophage
- Génome
- Capside
- Multiplication virale
- Concepts
- Virus
- Virus (Concepts)
- Fiche conceptuelle didactique